
BART는 Martin Uecker (Graz University of Technology), Jon Tamir (UT Austin), and Michael Lustig (UC Berkeley) 연구 그룹이 공동으로 개발한 MRI Reconstruction을 위한 Toolbox이다. 프로그래밍 라이브러리와 CLI 프로그램으로 구성되어 있으며, Parallel imaging과 compressed sensing에 사용할 수 있는 다차원 배열 연산, Fourier / wavelet 변환 등의 기능을 제공한다. (출처: 공식 홈페이지)
1. Install
참고: https://mrirecon.github.io/bart/installation.html
BART Toolbox
BART: Installation Figure: Simulated MRI images. Download Note: The software is intended for research use only and NOT FOR DIAGNOSTIC USE. It comes without any warranty (see LICENSE for details). Releases: It is recommended to download the latest release.
mrirecon.github.io
Window의 경우 위 링크에서 다운로드 받으면 되고,
Linux의 경우 command로 간단하게 설치 가능하다.
sudo apt-get install make gcc libfftw3-dev liblapacke-dev libpng-dev libopenblas-dev
wget https://github.com/mrirecon/bart/archive/v0.8.00.tar.gz
tar xzvf v0.8.00.tar.gz
cd bart-0.8.00
make
2022년 현재 기준으로 최신 버전은 0.8.00이고, 이전 버전을 다운받으려면 2~4번째 명령어에 다른 숫자를 넣어 주면 된다.
2. ESPIRiT을 이용한 coil sensitivity estimation
1) Setup
우선 Python에서 BART를 사용하기 위해 환경변수에 BART가 설치된 경로를 추가해 주어야 한다.
import sys, os
os.environ['TOOLBOX_PATH'] = "/path/to/bart-0.8.00"
os.environ['PATH'] = os.environ['TOOLBOX_PATH'] + ":" + os.environ['PATH']
sys.path.append(os.environ['TOOLBOX_PATH'] + "/python/")
from bart import bart
2) command 확인
BART의 ecalib command를 이용해 coil sensitivity estimation을 수행할 수 있다.

3) 데이터 준비
사용할 데이터는 다음과 같다. fastMRI multicoil knee 데이터셋을 이용했다.
참고: fastMRI 데이터셋 다운로드 및 사용법 Tutorial
fastMRI 데이터셋 다운로드 및 사용법 Tutorial
fastMRI는 Facebook AI와 NYU Langone Health가 2018년 공개한 MRI reconstruction을 위한 데이터셋이다. 공식 사이트: 데이터셋 개요와 file description, 리더보드를 확인할 수 있다. 데이터셋 다운로드: 데이터..
bo-10000.tistory.com
kspace = h5py.File(os.path.join(data_dir, fnames[0]))['kspace'][20]
import numpy as np
import matplotlib.pyplot as plt
def show_coils(data, slice_nums, cmap=None):
fig = plt.figure()
for i, num in enumerate(slice_nums):
plt.subplot(1, len(slice_nums), i+1)
plt.imshow(data[num], cmap=cmap)
print('kspace shape:', kspace.shape)
show_coils(np.log(np.abs(kspace)+1e-9), [0, 5, 10], cmap='gray')
def ifft2(img, norm='ortho'):
return np.fft.fftshift(np.fft.ifft2(np.fft.ifftshift(img), norm=norm))
show_coils(np.abs(ifft2(kspace)), [0, 5, 10], cmap='gray')
4) sensitivity map estimation
dimension을 바꿔 coil dimension이 맨 뒤로 가게 만들어주고, BART의 ecalib command를 이용해 sensitivity map estimation을 수행한다.
kspace = np.expand_dims(kspace.transpose(1, 2, 0), 0) #1, h, w, num_coils
sens_maps = bart(1, "ecalib -d0 -m1", kspace)
5) 결과 확인
print(sens_maps.shape)
print(sens_maps.dtype)
sens_maps = sens_maps[0].transpose(2, 0, 1) #num_coils, h, w
show_coils(np.abs(sens_maps), [0, 5, 10], cmap='gray')
위의 coil image와 결과가 일치하는 것을 확인할 수 있다.
전체를 plot하면 다음과 같다.
s_col = []
for i in range(3):
s_row = []
for j in range(5):
s_row.append(sens_maps[i*5+j])
s_col.append(np.concatenate(s_row, -1))
s_all = np.concatenate(s_col, -2)
plt.figure(figsize=(10, 10))
plt.imshow(np.abs(s_all), cmap='gray')
plt.axis('off');
6) Coil combine
생성한 coil sensitivity map을 이용해 각 coil image를 합쳐 보자.
recon_img = np.sum(sens_maps.conj()*ifft2(kspace), 0)
plt.figure(figsize=(7, 7))
plt.imshow(np.abs(recon_img), cmap='gray')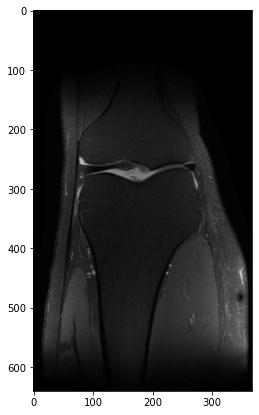
굿
참고한 사이트
'🔬 Medical Image > Processing' 카테고리의 다른 글
| TorchIO를 이용한 3D 이미지 transform (preprocessing/augmentation) (0) | 2022.06.07 |
|---|---|
| dicom 파일들을 nifti 파일로 바꿔주는 Python library 'dicom2nifti' (+일부 dicom 파일이 인식이 안되는 문제) (1) | 2021.12.21 |
| Pydicom library 이용해 dcm 파일 불러오기 (0) | 2021.08.04 |
| nibabel affine matrix의 의미와 affine transform 적용하는 방법 (0) | 2020.02.25 |